O teste RT-PCR (Real Time Polimerase Chain Reaction) constitui na resposta final e precisa sobre a presença do Sars-CoV-2, pertencente a família coronavidae, denominado também de novo coronavírus, na amostra analisada. Por ser considerada uma técnica molecular altamente sensível e específica, é considerado, desde o início da pandemia, a
metodologia padrão ouro para o diagnóstico do Covid-19.
O novo coronavírus é um vírus encapsulado não segmentado, com camada de lipídeo e 29 proteínas no seu capsídeo, com 50-200 nanômetros (0.05-0.2 micras) de diâmetro. Possui um genoma de RNA de fita simples, sentido positivo (+ssRNA) extremamente longo com
aproximadamente 30 mil nucleotídeos de comprimento distribuído em 4 bases: Guanina, Citosina, Adenina e Uracila.
O método padrão para o teste de coronavírus é por meio da reação em cadeia da polimerase amplamente utilizado na biologia molecular consistindo em amplificar, isto é produzir milhões a bilhões de cópias do material genético do vírus.
O RT-PCR, em tempo real, é utilizado para a detecção do novo coronavírus pela ampliação (aumento do número de cópias) da sequência alvo do gene Rdrp, gene E e gene N.
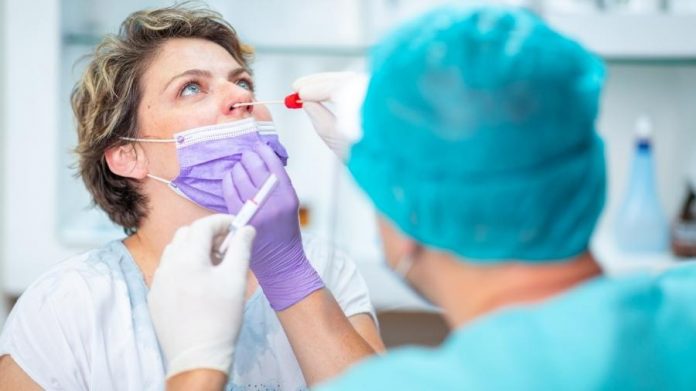
Entretanto, o desempenho analítico deste teste é altamente de pendente do procedimento de coleta das amostras o qual deve ser realizada por profissionais devidamente treinados munidos de EPIs , pois é sabido que grande parte do insucesso nas análises de RT-PCR se deve a erros de coleta.
A amostragem, feita com swab, é recomendada ser feita nas vias aéreas superiores (nasofaringe e/ou orofaringe) individualmente ou combinadas (neste último caso utilizam-se dois swabs). Após a coleta, o swab é imediatamente colocado em um tubo estéril contendo um meio de transporte viral.
Para detectar este vírus por PCR, as moléculas de RNA devem ser convertidas em sequências de DNA complementar através da transcriptase reversa de modo que o DNA recém formado possa ser amplificado via procedimento padrão da PCR. Esta abordagem é
reconhecida universalmente como RT-PCR.
Inicialmente é necessário extrair e isolar o RNA alvo contido na amostra. Isto é feito mediante a utilização de u m kit comercial Alguns procedimentos básicos são realizados para isolar e purificar o RNA : Na etapa de lise há a quebra da célula para expor o RNA; Em seguida, uma membrana de sílica retém e concentra o RNA. A etapa de lavagem consiste em quebrar e emulsionar a gordura e as proteínas que formam a membrana da célula normalmente feito utilizando soluções detergentes e por centrifugação. As etapas de lavagem removem estes resíduos contaminantes, restando apenas a membrana com o RNA. Por fim, na etapa de eluição, o eluente, tipo de solvente, interage com as amostras e
promove a separação dos componentes ocorre a liberação dos ácidos nucléicos (RNA) da membrana.
O próximo passo consiste na preparação das misturas reacionais para amplificação por meio da preparação de um a solução concentrada com o RNA purificado livre de proteínas, inibidores e outros contaminantes. Os seguintes ingredientes são adicionados à solução: a enzima transcriptase reversa, a enzima Taq DNA polimerase, dois primers, um íon de Magnésio e os nucleotídeos.
A Transcriptase reversa, também chamada de DNA polimerase RNA-dependente permite a transcrição da informação no sentido de RN A do virus , usado como um para a fabricação do DNA alvo ou do fragmento alvo a ser amplificado.
A Taq DNA Polimerase é a enzima capaz de utilizar uma fita molde de DNA para sintetizar (gerar) uma nova fita complementar. Esta enzima é extraída da bactéria Thermus aquaticus, que vive em água com temperatura de 75º C.
Os primers, também chamados de iniciadores, são pequenos fragmentos de DNA com cerca de 20 nucleotídeos, fita simples específicos e complementares às extremidades do DNA alvo que permite que a Taq DNA Polimerase dê continuidade ao processo de polimerização em cada fita.
O íon de Magnésio, bivalente, geralmente Cloretxo de Magnésio é utilizado como co-fator da Taq DNA Polimerase tendo como função a otimização de sua atividade.
Os nucleotídeos são as 4 bases nitrogenadas do DNA: Adenina, Timina, Citosina e Guanina adicionadas também à solução utilizados no processo de síntese das novas fitas.
Todos estes ingredientes são colocados em um tubo para que a PCR ocorra. O passo 1 é a desnaturação que ocorre à uma temperatura de 90-95ºC, isto é, nesta temperatura, a estrutura da dupla fita do DNA se desfaz com o rompimento d as pontes de Hidrogênio formando duas fitas simples.
Em seguida, com a temperat ura reduzida em torno de 58ºC, ocorre o anelamento , isto é os primers vão se anelar às extremidades do DNA alvo.
O terceiro passo (extensão), que ocorre em torno de 72ºC, a Taq DNA Polimerase que tem a capacidade de identificar esta estrutura dupla fita formada pela junção do seu DNA e do seu primer em decorrência do anelamento se associa ao DNA adicionando nucleotídeos que irão gerar as novas fitas.
Desta forma obtém-se duas fitas idênticas de DNA. O processo prossegue repetindo se em ciclos. Ao final do segundo ciclo se terá 4 moléculas, 8 no terceiro e assim por diante em progressão geométrica, sendo necessário cerca de 30 a 40 ciclos para que se obtenha o número das milhões de cópias necessárias.
Toda a PCR ocorre em um aparelho denominado Termociclador que tem como função variar as temperaturas ao longo dos ciclos de maneira automática.
Para monitorar a PCR em tempo real é necessário adicionar um ingrediente extra na forma de um agente fluorescente. Os níveis de fluorescência que aumentam de maneira proporcional ao número de cópias do alvo são traduzidos em informações digitais.
Para tanto, é necessário dispor de um aparelho detector das variações dos níveis de fluorescência e uma fonte de luz específica emitida por uma lâmpada halógena de tungstênio que vai excitar a fluorescência. Esta luz é recebida por uma câmara a qual converte a luz capturada em dados digitais.
Entretanto, o laudo com o resultado do teste fornecido ao paciente se limita a informar se foram ou não detectados os genes Rdrp, E e N do Sars-CoV-2 para detecção do novo coronavírus causador da Covid-19.
O laboratório alerta no laudo que um único resultado NÃO DETECTADO para Sars-CoV-2 não exclui o diagnóstico da Covid-19 e que vários fatores como coleta inadequada da amostra, tipo de amostra biológica, tempo decorrido entre a coleta e o início dos sintomas e oscilação da carga viral podem influenciar o resultado do exame. Sempre que houver discordância com o quadro clínico epidemiológico, o exame de RT qPCR deve ser repetido em outro tipo de amostra do trato respiratório respiratório
Fonte UNIMED
Mais informações: https://www.youtube.com/watch?v=ThG_02miq-4; https://www.youtube.com/watch?v=h2peadwu1zk
________________________________________________________________________

*José Eduardo W. de A. Cavalcanti
É engenheiro consultor, diretor do Departamento de Engenharia da Ambiental do Brasil, diretor da Divisão de Saneamento do Deinfra – Departamento de Infraestrutura da Federação das Indústrias do Estado de São Paulo (Fiesp), conselheiro do Instituto de Engenharia, e membro da Comissão Editorial da Revista Engenharia
E-mail: [email protected]
*Os artigos publicados com assinatura, não traduzem necessariamente a opinião do Instituto de Engenharia. Sua publicação obedece ao propósito de estimular o debate dos problemas brasileiros e de refletir as diversas tendências do pensamento contemporâneo